近日,西北大学生命科学学院孙士生教授团队在Advanced Science发表了题为“A Glycoproteome Data Mining Strategy for Characterizing Structural Features of Altered Glycans with Thymic Involution”的研究论文。该论文提出了一套适用于结构糖蛋白质组学深度分析的大数据挖掘策略,并应用于胸腺退化研究。该研究首次揭示了小鼠胸腺退化过程中N-糖基化的全景图谱和关键糖基化变化,为深入理解免疫衰老提供了全新的糖生物学视角。

胸腺是T细胞分化发育的核心器官,也是机体最早发生衰退的免疫器官,其结构及功能退化是免疫衰老的主要原因和特征之一。近年来,基于转录组学的胸腺衰老研究取得了诸多进展,但仍无法全面揭示胸腺衰老相关机制。糖链作为重要的信息通讯分子,在免疫识别和调控中发挥着重要作用。但是由于糖链结构的复杂性,位点和结构特征性的糖蛋白质组分析一直存在巨大技术挑战。近年来,随着各类完整糖肽分析软件的井喷式发展,大规模高通量的位点特异性糖链结构解析成为了可能。本研究充分利用孙士生教授团队自主开发的StrucGP软件结合TMT标记定量,借助产生的高深度、高分辨率的胸腺糖蛋白质组学定量数据,发展了一套高分辨率糖蛋白质组学数据的深度挖掘策略,实现了对复杂样品的整体和改变糖链结构特征及其调控网络的全面解析。
该研究提出的糖蛋白质组学数据挖掘策略主要包含四个核心模块:1)通过标记定量、糖肽预分离和质谱分析优化生成高质量的糖蛋白质组数据;2)通过系统的糖蛋白和糖链结构剖析全面表征复杂样品中的糖蛋白和糖链结构;3)通过多维度比较分析深度挖掘胸腺退化相关的特征糖链;4)借助多组学联合分析揭示糖基化改变参与的分子调控网络(图 1)。上述策略在小鼠胸腺中实现了18652种完整N-糖肽、5910个糖基化位点和3746种N-糖蛋白的定性定量分析,绘制了迄今最全面的胸腺糖蛋白质组图谱。
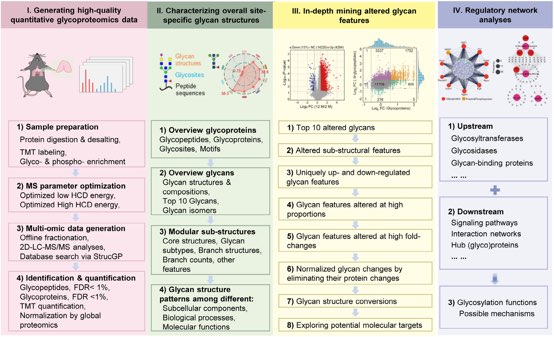
图1. 高质量N-糖蛋白质组数据获取和深度挖掘
通过深度数据挖掘,研究揭示了胸腺衰老过程中四种显著的糖链结构变化:1)LacdiNAc糖链显著升高,并主要富集于MHC-I类分子,提示其可能在抗原呈递过程中发挥重要作用;2)唾液酸化糖链(Neu5Ac/Neu5Gc)显著增加,主要参与多条免疫相关调控通路;3)平分型糖链(bisected GlcNAc)显著减少,同时伴随GlcNAc分支结构减少;4)同一糖基化位点上糖链结构呈现“重编程式”变化规律。此外,多组学整合分析还进一步揭示了差异糖基化相关的很多糖基转移酶、糖苷酶及糖结合蛋白等上游调控和互作因子,以及多条重要的下游信号通路(如PI3K-Akt、ECM受体互作及细胞周期调控)之间存在协同变化(图 2)。这些发现不仅充分展示了该数据挖掘策略的应用潜能,也为后续胸腺衰老机制研究提供了重要的糖生物学视角。
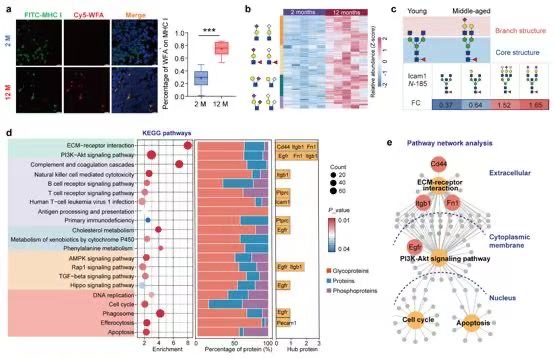
图2. 胸腺衰老过程中主要糖链结构变化及调控网络分析。a) LacdiNAc升高; b) 唾液酸(Neu5Ac和Neu5Gc)上调;c) 同一位点上的糖链结构重塑;d-e) 糖基化改变参与的信号通路(d)和通路网络(e)。
西北大学生命科学学院博士生张知达为该论文的第一作者,孙士生教授为通讯作者。该研究在国家自然科学基金、国家重点研发计划和陕西省重点项目等资助下完成。孙士生教授团队一直从事糖蛋白质组学方法学与应用研究,在糖链结构解析、软件开发、疾病糖蛋白质组学和肿瘤标志物发现研究等方面取得了多项重要研究成果,发表于Nature Biotechnology, Nature Methods, Nature Communications, Advanced Science, Redox Biology, Theranostics等期刊。
原文链接:advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202502013