基因表达对于决定细胞功能和可塑性至关重要,而不同的表观遗传修饰对于调控基因的转录水平起着关键作用。表观遗传调控是指在不改变DNA序列的前提下,通过改变DNA或染色质上的化学修饰来调控基因表达和遗传信息传递的过程。在表观遗传调控过程中,长链非编码RNA(lncRNA)、微小RNA(miRNA)等非编码RNA(ncRNA)发挥着关键作用,如lncRNA可以与组蛋白修饰相关的酶相互作用,实现位点特异性的转录调控,而miRNA可以直接结合特定的基因序列并沉默基因表达(Ha and Kim, 2014; Margueron and Reinberg, 2011)。因此,鉴定与特定染色质修饰相关的调控性RNA有助于我们进一步了解表观遗传调控机制。
尽管目前已有一些可用于探究与特定染色质修饰相关lncRNA的方法,如ChRIP-seq (Mondal et al., 2018)、CARIP-Seq (Kidder, 2018)、PIRCh-seq (Fang et al., 2019)和RT&Tag (Khyzha et al., 2022)等,但这些方法一方面基于化学或紫外交联过程,而交联会大大提高RNA与蛋白质的非特异性相互作用,导致鉴定到大量假阳性的RNA。同时,这些方法也均依赖高质量的ChIP级特异性抗体,而抗体制备过程中存在明显的批次效应,导致实验结果的重复性较差。因此,在表观遗传调控研究中,如何规避这些现有技术的限制是目前亟待解决的科学问题。

2024年7月31日,西北大学生命科学与医学部严健教授团队在Science Advances期刊上发表了题为Chrom-seq Identifies RNAs at Chromatin Marks的研究论文,报道了该研究组开发的一种名为Chrom-seq的新组学方法。Chrom-seq借助邻近标记技术,实验过程中无需交联和抗体,可以有效地捕获活细胞中与特定染色质修饰相互作用的RNA。具体来说,该技术是将工程化的染色质修饰识别结构域(eCRs)与工程化的抗坏血酸过氧化物酶(APEX2)相结合。同时,利用SunTag系统将eCR与10倍数量的APEX2融合,这样有效提高了RNA标记效率。APEX2可催化过氧化氢对生物素-苯胺(Btn-An)的氧化,在其附近产生大量携带氨基自由基(aminyl radical)的生物素-苯胺,它们半衰期短且具有高反应活性,因此可以与极为临近的RNA分子中的鸟苷(guanine)上C8碳原子反应形成共价化合物,从而标记这些RNA。借助于生物素,这些RNA可被凝集素(streptavidin)磁珠富集,进行后续的测序识别(图一)。
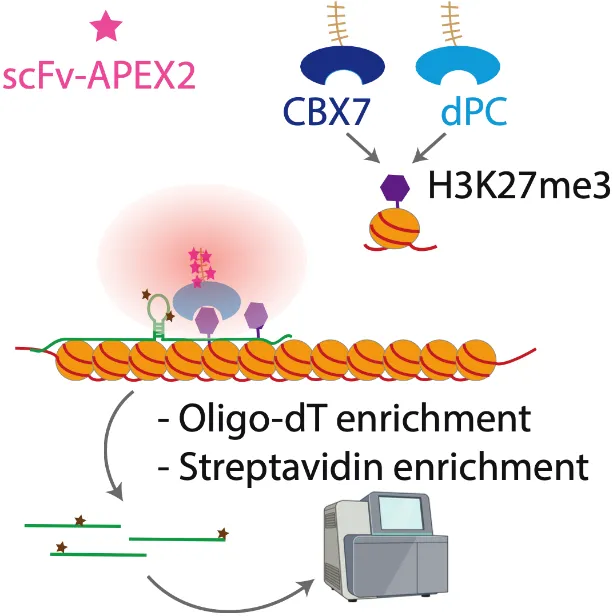
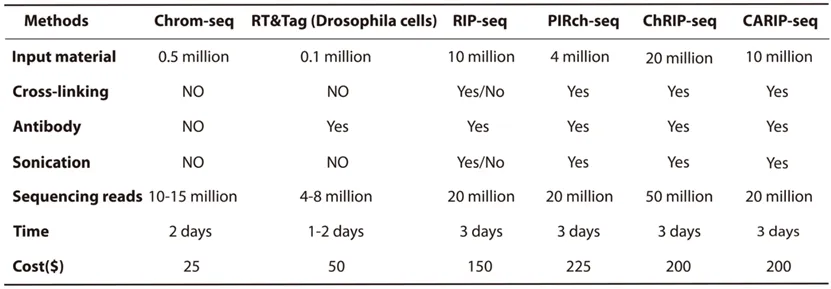
通过更换识别不同染色质修饰的eCR模块(如mCBX7/dPC可特异识别H3K27me3、mCBX1可特异识别H3K9me3和mTAF3可特异识别H3K4me3),团队在HEK293T细胞中利用Chrom-seq成功鉴定了大量与这些组蛋白修饰相关的RNA,并验证了部分RNA的表观调控功能。例如,团队首先聚焦于组蛋白H3K27me3修饰,鉴定到1437种与组蛋白H3K27me3修饰显著相关的RNA,包括许多先前已知的调控组蛋白H3K27me3修饰的RNA,如H19、NEAT1、MALAT1、TINCR和SOX2-OT等,证明了Chrom-seq方法的可行性和有效性。进一步的实验表明,SETD5-AS1和LINC00641这两个新发现的lncRNA在维持染色质特定区域的H3K27me3信号和周围基因表达中起着重要的作用。接着,研究团队又实用mCBX1模块对组蛋白H3K9me3修饰相关RNA进行了研究,鉴定到1301种与组蛋白H3K9me3修饰显著相关的RNA,如LINC00662和RP11-500G22.2等。通过进一步实验验证,发现敲低LINC00662会导致组蛋白H3K9me3信号的分布发生明显变化。有意思的是,尽管H3K27me3和H3K9me3修饰在基因组的分布中存在互斥,它们的关联RNA却有很大程度的重叠(~50%),暗示着它们之间存在着对话(cross-talk)和协同调控的可能性。最后,研究团队将Chrom-seq与其它现有方法进行比较,证实Chrom-seq在检测与染色质表观遗传修饰相关的RNA方面优于现有方法,如PIRCh-seq等。此外,Chrom-seq还具有效率高、成本低等优点(表一)。
表一. 比较Chrom-seq和其它基于抗体方法的优缺点。
总之,Chrom-seq为系统绘制与染色质表观遗传修饰相关的RNA提供了一种无需交联和抗体的高效新方法,为表观遗传调控的研究添置了新的工具,有助于我们进一步解析表观遗传调控机制。比如,我们可以利用Chrom-seq技术鉴定与肿瘤和衰老等相关的lncRNA,揭示这些lncRNA在调控肿瘤和衰老发生、发展过程中的调控机制。
严健为论文的独立通讯作者,西北大学樊立刚副教授及博士研究生孙伟为共同第一作者。香港城市大学生物医学系博士生吕薏潼、博士后巨富荣等也参与了该研究。此外,该项目也得到了国家自然科学基金、深圳市医学研究基金、香港研究资助局以及陕西省基础科学(化学与生物学)研究院的经费支持。
原文链接:
https://www.science.org/doi/10.1126/sciadv.adn1397
参考文献:
Fang, J., Ma, Q., Chu, C., Huang, B., Li, L., Cai, P., Batista, P.J., Tolentino, K.E.M., Xu, J., Li, R., et al. (2019). PIRCh-seq: functional classification of non-coding RNAs associated with distinct histone modifications. Genome Biol 20, 292.
Ha, M., and Kim, V.N. (2014). Regulation of microRNA biogenesis. Nat Rev Mol Cell Biol 15, 509-524.
Khyzha, N., Henikoff, S., and Ahmad, K. (2022). Profiling RNA at chromatin targets in situ by antibody-targeted tagmentation. Nat Methods 19, 1383-1392.
Kidder, B.L. (2018). CARIP-Seq and ChIP-Seq: Methods to Identify Chromatin-Associated RNAs and Protein-DNA Interactions in Embryonic Stem Cells. J Vis Exp.
Margueron, R., and Reinberg, D. (2011). The Polycomb complex PRC2 and its mark in life. Nature 469, 343-349.
Mondal, T., Subhash, S., and Kanduri, C. (2018). Chromatin RNA Immunoprecipitation (ChRIP). Methods Mol Biol 1689, 65-76.