近日,西北大学生命科学学院严健教授团队在《Journal of Translational Medicine》(Springer Nature旗下Q1期刊,影响因子7.5)发表题为“Targeting PURPL RNA enabled rejuvenation of senescence cells via epigenetic reprogramming”的研究论文。该研究首次揭示长链非编码RNA PURPL作为衰老过程中的关键表观遗传调控因子,通过调控染色质H3K9me3表观修饰驱动细胞衰老进程,为理解衰老机制及开发靶向干预策略提供了新靶点与理论依据。

细胞衰老是机体衰老及相关疾病发生发展的核心驱动因素,其特征包括p16/p21介导的细胞周期停滞、核纤层蛋白Lamin B1丢失以及组蛋白H3K9me3等表观遗传标记的改变。目前针对衰老细胞的清除策略仍存在特异性低、安全性风险高等局限。近年来研究表明,长链非编码RNA(lncRNA)作为基因表达与表观遗传调控的关键枢纽,在衰老进程中发挥重要作用。例如,靶向LINE-1 RNA可逆转衰老相关表观遗传标记,而抑制KCNQ1OT1则破环基因组稳定性。这些发现提示,靶向衰老相关lncRNA可能为延缓衰老及防治相关疾病提供新策略。
基于此,该研究构建了复制性衰老与药物诱导衰老两种细胞模型,通过转录组学分析系统筛选出一组在衰老过程中持续表达变化的长链非编码RNA,统称为衰老相关LncRNA。值得注意的是,在两种模型中PURPL均随衰老进程持续转录上调。进一步对人类组织表达数据库(GEO)的分析显示,PURPL在膀胱、乙状结肠等多种人体组织中的表达水平与年龄呈显著正相关,提示其潜在生物学重要性。
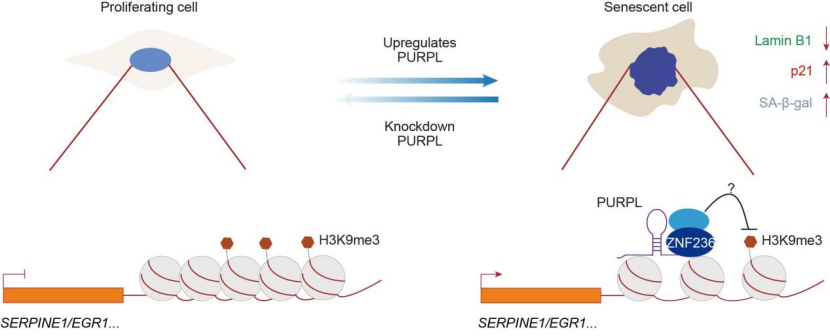
在功能验证中,研究团队利用CRISPRi技术在衰老细胞中敲低PURPL,成功逆转衰老表型,具体表现为SA-β-gal活性降低、p21表达下调及Lamin B1表达恢复,细胞形态趋于年轻化;相反,在年轻细胞中过表达PURPL则足以诱发典型衰老特征。该研究首次明确证实PURPL是调控细胞衰老状态的关键分子开关。
在机制层面,核质分离实验显示PURPL主要定位于细胞核。结合课题组前期发现其与H3K9me3修饰的相关性,本研究进一步发现在敲低或过表达PURPL后,细胞总H3K9me3水平发生显著改变。深入分析表明,PURPL缺失可特异性增强SERPINE1与EGR1等关键促衰老基因位点上的H3K9me3沉积,从而抑制其转录活性。通过CARPID技术筛选发现,PURPL能够与转录激活因子ZNF236及MTDH等发生特异性相互作用。这一结果提示,PURPL可能通过招募此类蛋白至特定基因组区域,介导局部H3K9me3擦除,进而解除对下游衰老相关基因的转录抑制,最终激活细胞衰老程序。
该研究系统揭示了PURPL作为表观遗传调控枢纽,通过介导H3K9me3重编程驱动细胞衰老的新型分子机制,不仅深化了对非编码RNA在衰老中作用机制的理解,更将PURPL确立为具有重要潜力的干预靶点,为发展针对年龄相关疾病的精准RNA疗法奠定了理论基础,同时为衰老干预策略开辟了新方向。
西北大学生命科学学院博士生王杰和硕士生杨晓(已毕业)、苏欣宇为共同第一作者,严健教授和宁永强博士为通讯作者。该工作得到国家自然科学基金面上项目、陕西省基础科学(化学、生物学)研究院重点项目、陕西省科技厅重点科技创新团队项目等资助支持。
全文链接: https://doi.org/10.1186/s12967-025-07208-5
PDF下载: https://rdcu.be/eLNsK
参考文献:
1. Di Micco R, Krizhanovsky V, Baker D, d'Adda di Fagagna F: Cellular senescence in ageing: from mechanisms to therapeutic opportunities. Nat Rev Mol Cell Biol 2021, 22:75-95.
2. Baker DJ, Wijshake T, Tchkonia T, LeBrasseur NK, Childs BG, van de Sluis B, Kirkland JL, van Deursen JM: Clearance of p16Ink4a-positive senescent cells delays ageing-associated disorders. Nature 2011, 479:232-236.
3. Lu Y, Brommer B, Tian X, Krishnan A, Meer M, Wang C, Vera DL, Zeng Q, Yu D, Bonkowski MS, et al: Reprogramming to recover youthful epigenetic information and restore vision. Nature 2020, 588:124-129.
4. Statello L, Guo CJ, Chen LL, Huarte M: Gene regulation by long non-coding RNAs and its biological functions. Nat Rev Mol Cell Biol 2021, 22:96-118.
5. Della Valle F, Reddy P, Yamamoto M, Liu P, Saera-Vila A, Bensaddek D, Zhang H, Prieto Martinez J, Abassi L, Celii M, et al: LINE-1 RNA causes heterochromatin erosion and is a target for amelioration of senescent phenotypes in progeroid syndromes. Sci Transl Med 2022, 14:eabl6057.
6. Fan L, Sun W, Lyu Y, Ju F, Sun W, Chen J, Ma H, Yang S, Zhou X, Wu N, et al: Chrom-seq identifies RNAs at chromatin marks. Sci Adv 2024, 10:eadn1397.
7.Yi W, Li J, Zhu X, Wang X, Fan L, Sun W, Liao L, Zhang J, Li X, Ye J, et al: CRISPR-assisted detection of RNA-protein interactions in living cells. Nat Methods 2020, 17:685-688.
8.Chen S, Chen Q, You X, Zhou Z, Kong N, Ambrosio F, Cao Y, Abdi R, Tao W: Using RNA therapeutics to promote healthy aging. Nat Aging 2025, 5:968-983.